Эпидемиология и Инфекционные болезни. Актуальные вопросы №1 / 2025
Цифровые решения (VGARus, SOLAR, «EpidSmart – модуль COVID») в системе эпидемиологического надзора за новой коронавирусной инфекцией (COVID-19)
Центральный НИИ эпидемиологии Роспотребнадзора, Москва, Россия
Цель исследования. Оценка, демонстрация эффективности и перспективы использования программных решений (цифровых платформ) в системе эпидемиологического надзора за инфекционными болезнями.
Материалы и методы. Обобщены сведения о функционировании программных решений в составе системы эпидемиологического надзора за период 2020–2023 гг. Проведен анализ сведений молекулярно-генетического мониторинга по данным платформы VGARus (Virus Genome Aggregator of Russia – платформа агрегирования результатов расшифровок генома возбудителей инфекционных и паразитарных заболеваний) за 2020–2023 гг., а также результатов ПЦР-исследований по данным платформы SOLAR (System of laboratory aggregation results – платформа агрегирования результатов лабораторных исследований) за 2022–2023 гг. и динамики заболеваемости новой коронавирусной инфекцией (COVID-19) населения Российской Федерации.
Результаты. Разработанные программные решения позволили эффективно модернизировать подсистемы эпидемиологического надзора за COVID-19 за счет изменений в сборе и хранении информации, а также при формировании эпидемиологического диагноза и краткосрочного прогноза. Представленные решения служат надежным источником обратной связи для повышения эффективности эпидемиологического контроля.
Заключение. Обосновано использование цифровых платформенных решений, созданных Центральным НИИ эпидемиологии Роспотребнадзора. Показана эффективность разработанных платформ: VGARus, выполняющей роль системы агрегации информации о геномах вирусов; SOLAR, позволяющей использовать данные результатов ПЦР-исследований для контроля за распространением возбудителя; «EpidSmart – модуль COVID», которая используется в качестве аналитического инструмента для проведения оперативного и ретроспективного эпидемиологического анализа и прогноза распространения генетических вариантов патогенов. Внедрение подобных программных решений является перспективным для совершенствования эпидемиологического надзора за другими инфекционными болезнями.
Значительный ущерб, нанесенный новой коронавирусной инфекцией (COVID-19), и масштаб распространения заболевания привели к необходимости максимально задействовать потенциал современной науки, а также инновационные разработки в области цифровых технологий для борьбы с пандемией. Возникла необходимость анализировать сведения о проявлениях эпидемического процесса новой коронавирусной инфекции, а также изменчивости возбудителя на всей территории Российской Федерации (РФ), что потребовало создания новых программных решений, помогающих выполнять эти задачи в оперативном режиме.
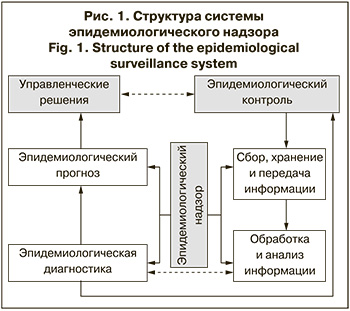
В классическом виде система эпидемиологического надзора за инфекционными болезнями на территории РФ представляет комплекс подсистем для принятия управленческих решений (рис. 1). Она является развитием идей В.Д. Белякова, В.И. Покровского и Б.Л. Черкасского о системе эпидемиологического надзора [1–3]. При работе по данной схеме осуществляются сбор, архивирование и передача информации, ее обработка и анализ, а также эпидемиологическая диагностика и разработка эпидемиологических прогнозов. Однако «в современных условиях рассматриваются два главных рычага интенсификации роста эффективности – ускорение научно-технического прогресса и совершенствование управления», – эти слова академика В.Д. Белякова о развитии системы противоэпидемических мероприятий актуальны и в настоящее время [1].
Для совершенствования эпидемиологического надзора за COVID-19 разработаны и внедрены в работу Роспотребнадзора платформа VGARus (Virus Genome Aggregator of Russia – платформа агрегирования результатов расшифровок генома возбудителей инфекционных и паразитарных заболеваний) – ключевой элемент системы молекулярно-генетического мониторинга за изменчивостью возбудителя, платформа SOLAR (System of Laboratory Aggregation Results – платформа агрегирования результатов лабораторных исследований) — централизованная база данных результатов ПЦР-исследований на РНК SARS-CoV-2 и «EpidSmart — модуль COVID» — аналитическая платформа для оперативного и ретроспективного эпидемиологического анализа с использованием больших данных (Big Data).
Цель исследования – оценка, демонстрация эффективности и перспективы использования программных решений (цифровых платформ) в системе эпидемиологического надзора за инфекционными болезнями.
Материалы и методы
Обобщен опыт использования и проведен анализ сценариев применения программных решений в составе системы эпидемиологического надзора за период 2020–2023 гг. на основе изучения рабочих процессов по анализу и прогнозу эпидемической обстановки по новой коронавирусной инфекции, а также научных публикаций.
Молекулярно-генетический мониторинг изменчивости возбудителя COVID-19 за период 2021–2023 гг. с учетом сведений эпидемиологического анамнеза осуществлен с использованием данных платформы VGARus.
Анализ сведений о результатах ПЦР-исследований РНК SARS-CoV-2 за период 2022–2023 гг. проведен по данным платформы SOLAR.
Динамика заболеваемости COVID-19 населения субъектов РФ за аналогичный период проанализирована по данным официальной отчетной формы «Мониторинг о количестве заболевших коронавирусной инфекцией».
Корреляционный анализ между динамикой заболеваемости населения COVID-19 и регистрацией положительных ПЦР-результатов на РНК SARS-CoV-2, а также между динамикой заболеваемости населения COVID-19 и долей положительных ПЦР-результатов в субъектах РФ проведен за период 2022–2023 гг. Для определения взаимосвязи использован коэффициент корреляции Пирсона.
Оценка статистической значимости полученного значения коэффициента корреляции проведена с использованием метода Монте–Карло. Для оценки причинности использован метод Грэнджера.
Статистическую обработку результатов выполняли с использованием аналитических инструментов SciPy [4], Pandas [5], Statsmodels [6].
Результаты
Платформа VGARus
Данная цифровая платформа создана в ФБУН «Центральный НИИ эпидемиологии» Роспотребнадзора (далее – ЦНИИЭ) для осуществления мониторинга изменчивости SARS-CoV-2. По состоянию на 1 августа 2024 г. на платформу загружено более 327 тыс. нуклеотидных последовательностей. Из них более 206 800 представляют собой полные геномы, 120 180 – фрагменты генома.
Представлены генетические последовательности вариантов SARS-CoV-2: Alpha – более 1600, Beta – более 140, Delta – более 62 300, Gamma – более 20, Omicron – более 232 900. Метаданные платформы включают в себя 20 различных показателей, в том числе данные эпидемиологического анамнеза, и имеют обезличенный характер.
К платформе подключено более 130 организаций, представляющих различные ведомства. Все загруженные последовательности обрабатываются с помощью программного обеспечения, встроенного в платформу, которое определяет геновариант возбудителя в автоматическом режиме. Результаты становятся доступными для анал...












