Эпидемиология и Инфекционные болезни. Актуальные вопросы №3 / 2014
Разработка и апробация ДНК-чипа для индикации возбудителей особо опасных инфекций
Центральный НИИ эпидемиологии Роспотребнадзора, Москва; НИИ медицины труда РАН, Москва; Российский научно-исследовательский противочумный институт «Микроб» Роспотребнадзора, Саратов
Постоянная необходимость мониторинга за особо опасными инфекционными заболеваниями обусловливает актуальность разработки новых эффективных методов идентификации возбудителей этих заболеваний.
Цель исследования. Создание и апробация набора реагентов, включающего ДНК-чип, для выявления возбудителей сибирской язвы, чумы, туляремии, холеры, легионеллеза и бруцеллеза.
Материалы и методы. Олигонуклеотидные зонды, несущие аминогруппы на 5,-конце, наносились на поверхность слайдов с альдегидным покрытием с помощью плоттера для бесконтактной печати. После выделения ДНК из анактивированных культур проводили стадию мультипраймерной ПЦР, затем стадию транскрипции с включением РНК-детектирующей метки. Продукты транскрипции гибридизовали на ДНК-чипе, затем с помощью лазерного сканера проводили анализ флюоресцентных профилей.
Результаты. Разработан набор реагентов, включающих ДНК-чип, для выявления возбудителей особо опасных инфекционных заболеваний, позволяющий провести анализ 48 образцов. Время анализа составляет 5,5 ч.
Заключение. Набор реагентов обладает чувствительностью и специфичностью, что дает возможность применять его я в лабораториях регионального уровня и референсных центрах.
Для осуществления мониторинга за особо опасными инфекциями и проведения своевременных противоэпидемических мероприятий актуальной является разработка новых эффективных диагностических наборов. На сегодняшний день все большее распространение получают методы индикации и идентификации возбудителей различных инфекций с использованием ДНК-чипов. Особенностью данной технологии является возможность проведения одномоментного мультилокусного анализа за сравнительно короткое время при высоких уровнях чувствительности и специфичности. Наравне с полимеразной цепной реакцией (ПЦР) ДНК-чипы могут найти применение для идентификации и генетического типирования возбудителей особо опасных инфекционных заболеваний в профильных лабораториях территориального, регионального и федерального уровней.
Целью настоящей работы являлось конструирование и апробация на панели референтных штаммов микроорганизмов набора реагентов, включающего ДНК-чип, для дифференциального выявления возбудителей особо опасных инфекций: сибирской язвы – Bacillus anthracis, чумы – Yersinia pestis, туляремии – Francisella tularensis, холеры – Vibrio cholerae, легионеллеза – Legionella pneumophila, бруцеллеза – Brucella abortus. Выбор данных микроорганизмов для диагностики с помощью ДНК-чипа обусловлен, прежде всего, высокой вероятностью использования их в качестве средств биотерроризма [1, 2].
Материалы и методы
 Выбор структур олигонуклеотидных праймеров и зондов проводили с использованием программного пакета Vector NTI («Invitrogen Corporation», США), а также ресурса, предлагаемого на серверe http://www.basic.northwestern.edu/biotools/oligocalc.html. Все структуры зондов и праймеров сравнивались с помощью программы BLAST (информационный ресурс NCBI) с последовательностями ДНК, депонированными в базе данных GenBank. Олигонуклеотдиные зодны и праймеры были синтезированы в ФБУН «Центральный НИИ эпидемиологии» Роспотребнадзора, Москва (далее – ЦНИИЭ). Зонды, предназначенные для последующей иммобилизации на слайдах, содержали аминогруппу на 5’-конце. В состав одного из праймеров каждой пары был включен сайт узнавания для ДНК-зависимой РНК-полимеразы бактериофага Т7. Структуры олигонуклеотидных праймеров и зондов представлены в табл. 1.
Выбор структур олигонуклеотидных праймеров и зондов проводили с использованием программного пакета Vector NTI («Invitrogen Corporation», США), а также ресурса, предлагаемого на серверe http://www.basic.northwestern.edu/biotools/oligocalc.html. Все структуры зондов и праймеров сравнивались с помощью программы BLAST (информационный ресурс NCBI) с последовательностями ДНК, депонированными в базе данных GenBank. Олигонуклеотдиные зодны и праймеры были синтезированы в ФБУН «Центральный НИИ эпидемиологии» Роспотребнадзора, Москва (далее – ЦНИИЭ). Зонды, предназначенные для последующей иммобилизации на слайдах, содержали аминогруппу на 5’-конце. В состав одного из праймеров каждой пары был включен сайт узнавания для ДНК-зависимой РНК-полимеразы бактериофага Т7. Структуры олигонуклеотидных праймеров и зондов представлены в табл. 1.
Производство ДНК-чипов
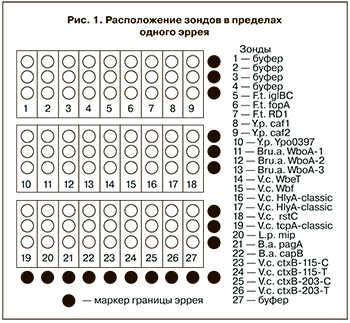
Препараты олигонуклеотидных зондов наносили на микроскопные слайды с поверхностью, модифицированной альдегидными группами, VALS («СЕL Associates Inc.», США) методом бесконтактной печати с помощью пьезоэлектрического робота для микропечати S3 («Scienion AG», Германия) в виде индивидуальных пятен (спотов) диаметром 350 мкм в трех повторах. Олигонуклеотидные зонды и контрольные споты были нанесены в пределах каждой из двенадцати зон (эрреев) на слайде идентичным образом (рис. 1). Далее слайды инкубировали в течение ночи при комнатной температуре, обрабатывали свежеприготовленным 0,25% раствором боргидрида натрия, промывали 0,2% раствором додецилсульфата натрия (SDS), затем дистиллированной водой и высушивали.
Материал для исследования
Для изучения аналитической чувствительности и специфичности ДНК-чипа были использованы штаммы следующих микроорганизмов: Bacillus anthracis (13 штаммов), Yersinia pestis (8), Francisella tularensis (9), Vibrio choleraе (9), Legionella pneumophilla (3), Brucella abortus (1), Bacillus cereus (1), Bacillus subtilis (1), Yersinia psedotuberculosis (1), Yersinia enterocolitica (1), Escherichia coli (2), Vibrio parahaemolyticus (1), Vibrio vulnificus (1), Vibrio mimicus (1). Данные штаммы были предоставлены ФБУН «Государственный научный центр прикладной микробиологии и биотехнологии» Роспотребнадзора (Оболенск) и ФКУЗ Российский научно-исследовательский противочумный институт «Микроб» (Саратов; далее – РосНИПЧИ «Микроб»). Межлабораторные испытания ДНК-чипа проводили в РосНИПЧИ «Микроб» на панелях штаммов, включавших 8 штаммов B. anthracis, по одному штамму B. cereus, B. subtilis и megatherium, 7 штаммов Y. pestis, 1 штамм Y. enterocolitica, 1 штамм Y. psedotuberculosis, 8 штаммов F. tularensis, по одному штамму B. abortus, B. melitensis, B. suis, B. ovis, B. canis и B. neotomae, 10 штаммов V. cholerae, по одному штамму V. parahaemolyticus, V. mimicus и V. vulnificus, 3 штамма L. pneumophila и...












